
ภาพรวมของการวิเคราะห์นิวคลีโอไทด์โดยใช้เทคโนโลยี next generation sequencing
Sanger sequencing เป็นวิธีการหาลำดับนิวคลีโอไทด์รุ่นแรกที่ถูกพัฒนาโดย Frederick Sanger ในปีพ.ศ. 2515 วิธีการนี้เรียกหนึ่งอีกอย่างว่า การจัดลำดับนิวคลีโอไทด์ตามการสิ้นสุดของโซ่ (dideoxy chain termination) วิธีนี้มีการใช้กันอย่างแพร่หลายเพื่อการหาลำดับดีเอ็นเอ เนื่องจากมีความแม่นยำมากกว่าร้อยละ 99.9 แต่อย่างไรก็ตาม กลับพบว่าวิธีการนี้ยังไม่มีประสิทธิภาพเท่าที่ควร เนื่องจาก Sanger sequencing สามารถอ่านลำดับเบสได้เพียง 1,000 - 1,200 เบส และตรวจหาลำดับนิวคลีโอไทด์ได้ครั้งละหนึ่งตัวอย่างเท่านั้น จึงทำให้มีราคาต่อตัวอย่างค่อนข้างแพง ดังนั้นหลายบริษัทจึงได้มีการพัฒนาเทคโนโลยีในการหาลำดับนิวคลีโอไทด์ในปริมาณมากขึ้นมา ซึ่งเรียกว่า New Generation Sequencing (NGS) โดยมีจุดประสงค์เพื่อลดต้นทุนค่าใช้จ่ายลงและยังสามารถทำได้หลายตัวอย่างต่อการทำงานของเครื่องหนึ่งครั้ง Roche 454 GS-20 เป็นระบบแพลตฟอร์มแรกของ NGS ที่ถูกคิดค้นและพัฒนาออกสู่ตลาดในช่วงปีพ.ศ. 2548 โดยสามารถอ่านลำดับนิวคลีโอไทด์ได้มากถึง 20 ล้านเบสในเวลาเดียวกัน และในช่วงระยะเวลา 15 ปีที่ผ่านมา เทคโนโลยี NGS ได้มีการพัฒนาขึ้นมาในหลากหลายรูปแบบ ดังแสดงในรูป
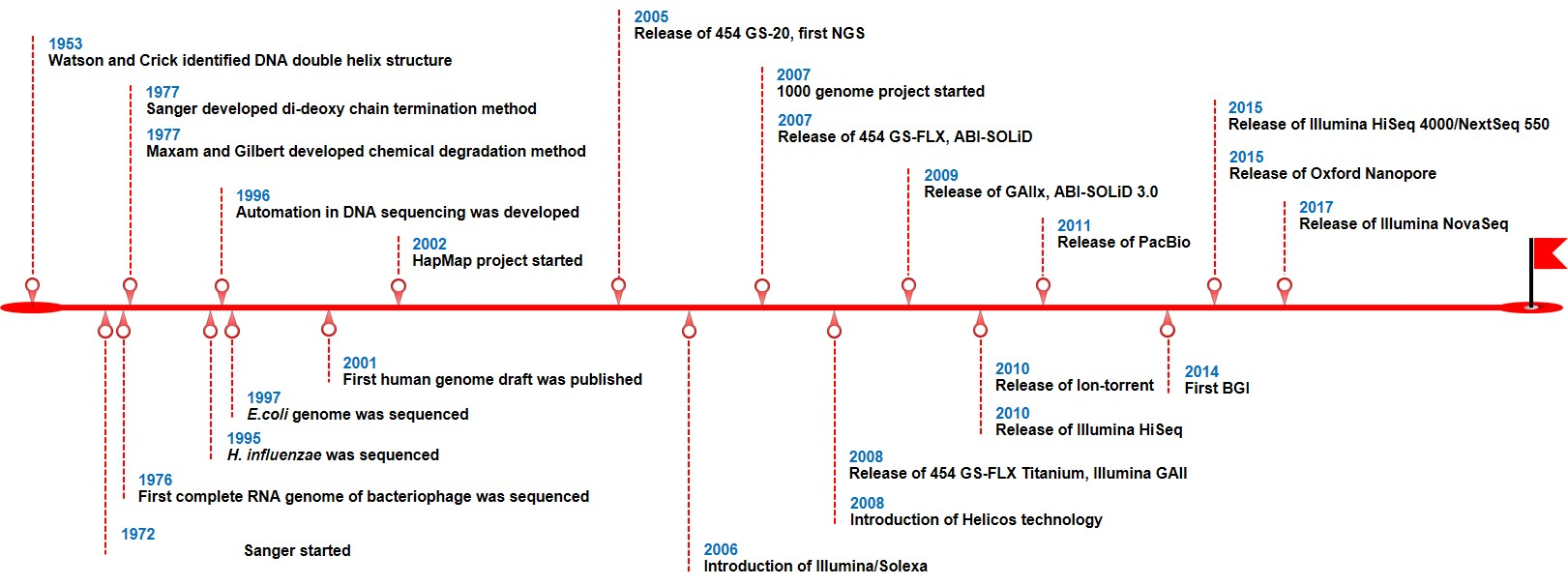
ในปัจจุบันเทคโนโลยี NGS กำลังเป็นที่นิยมและใช้กันอย่างแพร่หลายทั่วโลก เนื่องจากสามารถสร้างข้อมูลลำดับเบสที่มีขนาดใหญ่ (มากกว่า 1,000,000 เบส) ได้อย่างรวดเร็วในคราวเดียว โดยจะใช้วิธีการเพิ่มจำนวนดีเอ็นเอไปพร้อมๆ กับการอ่านลำดับเบส เทคโนโลยี NGS นี้ประกอบด้วย แพลตฟอร์ม Roche 454 Illumina และ ABI-SOLiD ซึ่งถือเป็นเทคโนโลยีที่สำคัญ เพื่อช่วยในการวินิจฉัยโรค ศึกษาหาลำดับนิวคลีโอไทด์ของสารพันธุกรรมของสิ่งมีชีวิตที่สนใจ และวิเคราะห์เชิงวิวัฒนาการของพืชและสัตว์
References :
Gupta AK, Gupta U. Next generation sequencing and its applications. Anim Biotechnol 2020:395–421. doi:10.1016/b978-0-12-811710-1.00018-5